Total Tayangan Halaman
Kamis, 09 September 2021
Jumat, 03 September 2021
REPLIKASI DNA
REPLIKASI DNA
Replikasi DNA adalah suatu proses penggandaan DNA sebagai materi genetik makhluk hidup. Proses ini sangat penting dalam tahapan perkembangbiakan atau pembelahan sel (fase S siklus sel). Materi DNA yang telah digandakan kemudian akan dibagi ke masing-masing anakan sel yang baru. Pada artikel ini kita akan membahas bagaimana proses mekanisme replikasi ini berlangsung.
Struktur Dasar dan Pasangan Basa DNA
Kita ketahui bahwa struktur DNA berupa double helix yang tediri dari dua untai rantai polimer DNA yang saling berpasangan secara komplementer. Struktur dasar tersebut dapat disimak di gambar di bawah ini:

Struktur dan pasangan basa komplementer tersebut penting untuk diperthatikan dalam mempelajari mekanisme replikasi DNA. Adapun untuk susunan komplementer dari basa DNA terdiri dari pasangan satu basa purin dengan satu basa pirimidin. Basa purin terdiri dari adenine (A) dan guanine (G) sedangkan pirimidin terdiri dari cysotine (C) dan thymine (T). Pada RNA, thymine diganti oleh uracyl (U).
Dalam berpasangan, adenine (A) berpasangan dengan thymine (T) sedangkan guanine (G) berpasangan dengan cytosine (C). Adapun terbentuknya pasangan tersebut dikarenakan adanya ikatan hidrogen antara kedua jenis basa. Ikatan hidrogen ini dapat kita simak di gambar di bawah ini:

Mengetahui susunan dan pasangan basa DNA ini penting untuk mempelajari bagaimana DNA diduplikasi atau digandakan.
Template dan Penyalinan DNA
Dasar dari penyalinan DNA adalah penyocokan pasangan basa DNA dari template atau DNA induk. Pada tahap awal, struktur double helix akan dipisahkan dan masing-masing untaian tunggal akan menjadi template atau DNA induk. Basa G akan berpasangan dengan basa C yang baru dan basa T hanya akan berpasangan dengan basa A yang baru. Dengan demikian, masing-masing helai DNA akan menjadi dasar salinan template untuk dibentuknya helai komplementer DNA yang baru.

Proses pembentukan untaian baru DNA atau polimerisasi DNA dijalankan oleh enzim yang dinamakan DNA polimerase. Enzim ini ditemukan tahun 1957. DNA polimerase menggunakan deoksiribonukleosida trifosfat sebagai substratnya dan kemudian menggabungkan atau mempolimerisasi substrat tersebut menjadi untaian DNA baru. Adapun sebagai dasar cetaknya, cukup membutuhkan satu untaian dari template DNA induk.

Proses polimerisasi DNA oleh DNA polimerase dimulai dari datangnya deoksinukleosida trifosfat bebas yang sesuai dengan pasangan basa dari ujung 3′ DNA template. Saat deoksinukleosida trifosfat ini menempel, bentuk DNA polimerase yang seperti tangan akan berubah, seperti tangan yang mengeratkan genggamannya ke DNA. Akibat perubahan ini, enzim mendekatkan ujung gugus OH di posisi 3′ ke gugus firofiosfat pada deoksinukleosida trifosfat. Hal ini menyebabkan reaksi, dibentuknya ikatan gula fosfat dan sebagai hasil samping keluar gugus firofosfat. Proses ini dapat disimak pada bagan di bawah ini:

Adapun yang dimaksud dengan 3′ dan 5′ adalah sistem penomoran atau posisi dari atom karbon pada cincin deoksiribosa pada DNA atau ribosa pada RNA. Penomoran ini dapat dilihat pada gambar di bawah ini:

Proses Replikasi Asimetris pada Garpu Replikasi DNA
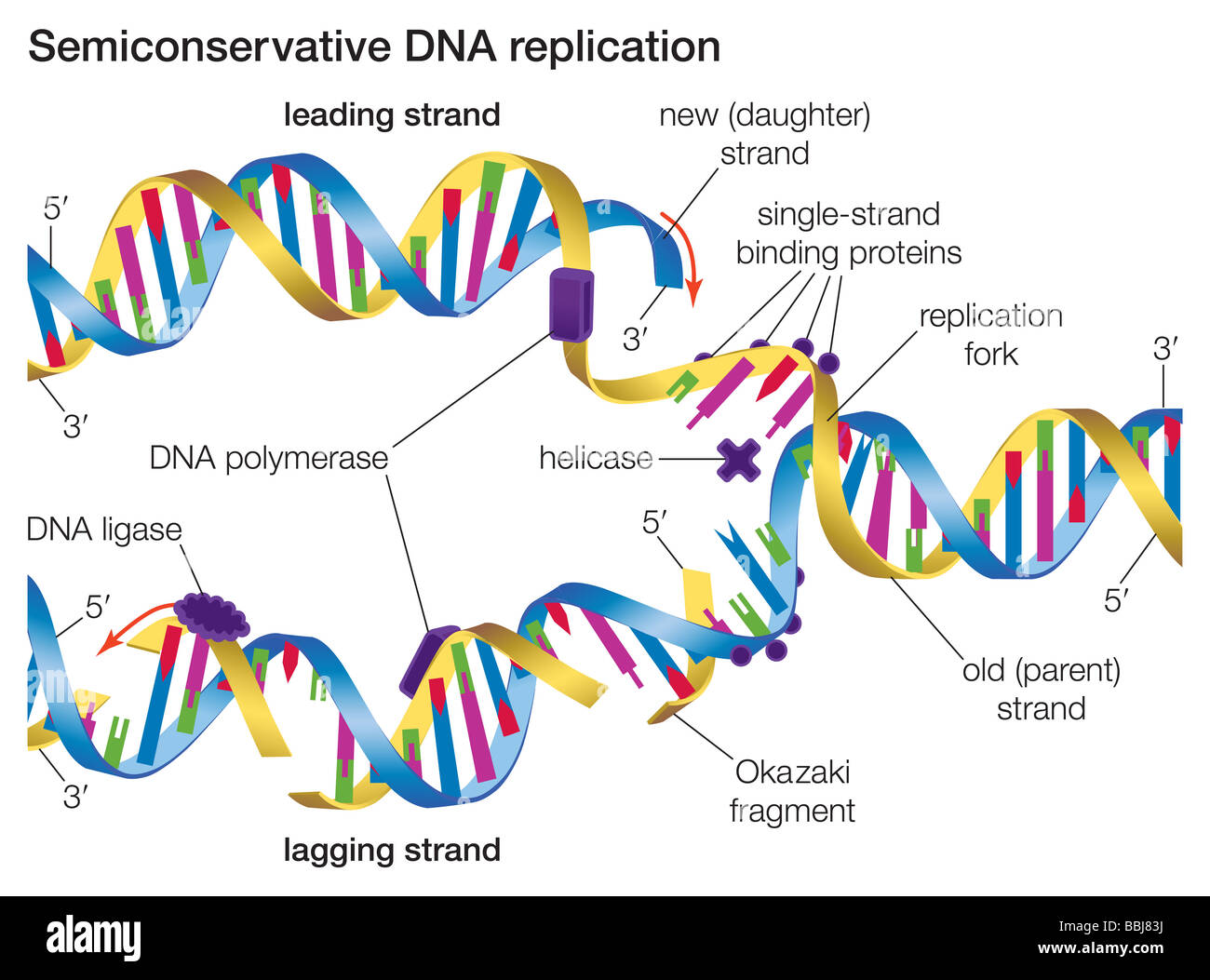
Proses replikasi DNA membutuhkan penguraian terlebih dahulu bentuk double helix. Akan tetapi, proses replikasi DNA tidak perlu menunggu struktur double helix ini dibuka seluruhnya. Ketika sebagian dari DNA mulai terbuka, maka proses replikasi ini dapat dimulai. Apabila kita bayangkan, maka bentuk ini seperti “garpu”. Tampak struktur proses replikasi DNA seperti gambar di bawah ini:

Dari gambar di atas, tampak bahwa kedua helai untaian DNA induk menjadi template bagi terbentuknya DNA yang baru. Proses di atas dikatakan bahwa replikasi DNA berlangsung secara “semikonservatif“.
Di garpu replikasi inilah tempat terjadinya kegiatan utama proses replikasi. Pada saat proses replikasi terjadi, di titik tersebut tersusun kompleks multiprotein dan multienzim yang terlibat dalam proses tersebut.
Apabila kita perhatikan di gambar sebelumnya, kita ketahui bahwa arah replikasi terjadi dari 5′ ke 3′ dari template yang berorientasi 3′ ke 5′. Namun, saat double helix dibuka, kedua untaian memiliki orientasi yang berbeda satu sama lain (antiparalel). Hal ini menyebabkan proses replikasi berbeda untuk setiap helai DNA dan juga membutuhkan set protein dan enzim yang berbeda pula. Oleh sebab itu, proses replikasi di garpu replikasi bukanlah proses yang simetris (asimetris).
Leading Strand, Lagging Strand, dan Fragmen Okazaki
Di garpu replikasi, helai DNA dengan orientasi 3′ ke 5′ akan diproses secara kontinu. Saat fragmen dibuka sedikit demi sedikit, enzim polimerase akan memproses dari satu basa ke basa selanjutnya. Helai DNA yang orientasi ini disebut leading strand. Adapun helai DNA dengan orientasi yang berlawanan disebut lagging strand.
Adapun pada helai DNA yang berlawanan, proses ini tidak bisa dilakukan secara kontinu. Bagaimana cara replikasi DNA di template dengan orientasi 5′ ke 3′? Pada tahun 1960-an, dengan memanfaatkan zat radioaktif 3H-thymidine peneliti menemukan terdapat potongan-potongan DNA di lagging strand dengan panjang 1000-2000 nukleotida yang dinamakan fragmen Okazaki. Fragmen Okazaki juga disintesis dengan arah 5′ ke 3′ dan kemudian disambungkan setelah potongan-potongan ini disintesis. Untuk lebih jelas, lihat gambar di bawah ini:

Perlunya Sistem Proofreading pada Replikasi DNA
Proses replikasi DNA berlangsung relatif sangat cepat. Akan tetapi, walaupun cepat, namun memiliki tingkat ketepatan yang baik. Tingkat kesalahan pada manusia hanya satu kesalahan diantara 109 nukleotida yang disalin. Bayangkan seorang sekretaris hanya memiliki tingkat kesalahan satu huruf diantara 1 milyar huruf yang dia ketik. Tingkat ketepatan tinggi ini dikarenakan adanya sistem proofreading di tingkat seluler.
Apabila kita perhatikan, ternyata sistem pasangan basa cukup rentan mengalami kesalahan. Jika ada sedikit perubahan geometri double helix, dapat terjadi dua ikatan hidrogen antara G dan T. Padahal seharusnya G dan T tidak berpasangan (G dengan C dan A dengan T). Selain itu, dapat terbentuk bentuk tautomeric yang langka dari keempat basa ini secara sementara
Perubahan tautomeric adalah berpindahnya atom hidrogen di dalam satu molekul sehingga merubah karakter dan bentuk isomer dari molekul tersebut. Perubahan tautomeric ini dapat memungkinkan C berpasangan dengan A dari apda G. Bentuk tautomeric ini terdapat pada 1 diantara 104 sampai 105 molekul basa. Untuk lebih jelas, dapat disimak di gambar di bawah ini:

DNA Polimerase Berperan dalam Proofreading dan Editing DNA
DNA polimerase merupakan lini pertama sistem proofreading saat replikasi berlangsung. Selain melakukan replikasi, DNA polimerase juga dapat berfungsi mengecek dan mengedit DNA yang salah. Hal ini diketahui dari studi atau penelitian terhadap berbagai macam DNA polimerase seperti pada bakteri dan bahkan virus.

Seperti dijelaskan di atas, terkadang dapat terjadi nukleotida yang salah dimasukan ke dalam strand DNA yang baru. Dalam keadaan normal, nukleotida yang tepat membuat proses replikasi berjalan secara lancar. Namun pada saat masuk nukleotida yang salah, proses ini berjalan lebih jauh lebih lambat. Hal ini memungkinkan enzim untuk melakukan cek ulang apakah nukleotida yang dimasukan sudah sesuai atau sebelum dilakukan proses pembentukan ikatan kovalen. Jadi cara ini berlaku sebelum terjadi pembentukan ikatan kovalen fosfodiester nukleotida yang baru.
Sistem berikutnya adalah exonuclease proofreading. Proses ini berlangsung segera setelah ikatan kovalen dengan nukleotida yang salah terbentuk. Dikarenakan adanya ketidakcocokan, maka ikatan hidrogen tidak terbentuk dengan semestinya. Hal ini menyebabkan posisi molekul tidak sesuai sehingga DNA polimerase akan berhenti melakukan replikasi.

Nukleotida yang salah ini kemudian akan dipindahkan ke situs lain dalam enzim DNA polimerase. Kemudian, nukleotida yang salah ini akan dibuang dan diganti dengan nukleotida yang seharusnya.
Apabila dibandingkan dengan proses translasi RNA, kesalahan pada proses replikasi 100.000 kali lebih sedikit. Hal ini berkat adanya sistem proofreading tersebut. RNA polimerase tidak memiliki kemampuan editing seperti halnya DNA polimerase.
Sintesis RNA Primer Pendek pada Template Laging Strand DNA
Di leading strand, arah replikasi searah dengan pembukaan struktur double helix. Oleh sebab itu, hanya diperlukan satu kali primer khusus yaitu pada saat dimulainya replikasi agar DNA polimerase bisa mulai bekerja. Hal ini berbeda dengan lagging strand. Dijelaskan sebelumnya bahwa pada lagging strand, arah replikasi berlawanan dengan arah pembukaan double helix. Saat DNA polimerase selesai membuat satu fragmen Okazaki, maka enzim harus kembali ke garpu replikasi untuk membuat fragmen Okazaki yang baru. Untuk setiap dimulainya fragmen Okazaki baru perlu ada primer khusus untuk memulai replikasi.
Untuk keperluan ini, ada enzim khusus yang dinamakan DNA primase. Enzim ini berfungsi membuat primer di lagging strand. Primer yang dibentuk berupa segmen RNA pendek yang dikenali oleh DNA polimerase untuk memulai replikasi fragmen Okazaki yang baru. Pada eukariota, RNA pendek itu terdiri dari 10 untai nukleotida dan dibuat dalam interval 100-200 nukleotida.

Proses Penggabungan Fragmen Okazaki
Fragmen Okazaki akan selesai dibentuk apabila ujung 5′ bertemu dengan ujing 3′ dari RNA primer fragmen sebelumnya. Setelahnya maka lagging strand akan terdiri dari RNA primer dan fragmen Okazaki. Dibutuhkan proses lanjutan berupa penghilangan RNA primer dan menyambungkan fragmen Okazaki menjadi satu strand yang utuh.

Proses ini dijalankan oleh satu sistem perbaikan DNA khusus yang menghilangkan RNA primer dan digantikan dengan DNA. Setelah itu, fragmen yang masih terputus akan disambungkan oleh DNA ligase (reaksi di gambar di bawah).

Kenapa harus dibutuhkan RNA primer? Kenapa tidak langsung DNA primer saja sehingga tidak perlu dihapus atau tidak perlu primer sama sekali? Ternyata hal ini berkaitan untuk mengurangi jumlah eror atau kesalahan saat replikasi DNA. Enzim DNA polimerase yang dapat memulai replikasi tanpa primer ternyata tidak efisien dalam proses koreksi DNA. Dengan menggunakan RNA primer, tubuh secara otomatis menandakan tempat replikasi yang rentan eror sehingga diperlukan perlakuan khusus agar proses replikasi dapat berlangsung seakurat mungkin.
Protein Pembuka Struktur Double Helix
Struktur double helix mencegah baik proses replikasi maupun translasi dari gen. Sebab itu, maka sebelum proses replikasi, maka struktur ini harus dibuka terlebih dahulu. Agar DNA tetap dibuka, maka ada dua protein yang bereperan yaitu DNA helikase dan single-strand DNA-binding protein.
DNA Helikase
DNA helikase pertama kali diisolasi sebagai enzim yang menghidrolisis ATP saat menempel ke DNA helai tunggal (single strand). Dengan melakukan hidrolisis ATP, DNA helikase dapat menempel dan bergerak sepanjang DNA dan membelah struktur double helix DNA menjadi dua single strand DNA.

Berbeda dengan RNA polimerase yang hanya bisa berjalan dari arah template 5′ ke 3′, DNA helikase dapat berjalan dua arah. DNA helikase yang berjalan dari 5′ ke 3′ pada lagging strand biasanya mengambil fungsi predominan dalam membelah struktur double helix DNA.
Single-Strand DNA-Binding (SSB) Protein

Protein yang kedua adalah single-strand DNA-binding (SSB) protein atau nama lainnya helix-destabilizing protein. Protein ini berfungsi menstabilkan sruktur single strand DNA. DNA yang telah dibelah dari struktur double helix dapat membentuk gulungan yang mengganggu proses replikasi. Dengan adanya SSB, single-strand DNA dapat tetap lurus sehingga memudahkan proses replikasi.

Struktur Sliding Ring Menahan DNA Polimerase menempel ke DNA Template Saat Proses Replikasi
Sebenarnya dalam kondisi bebas, DNA polimerase hanya dapat mensintesis potongan pendek DNA sebelum kemudian terlepas dari DNA template. Kecenderungan sifat DNA polimerase untuk lepas ini penting untuk memungkinkan DNA polimerase yang baru saja menyelesaikan fragmen Okazaki di lagging strand untuk lepas dan menempel ke tempat yang baru.
Akan tetapi pada leading strand, tentu sifat disosiasi ini tidak menguntungkan sehingga diperlukan protein lain untuk membantu DNA polimerase tetap menempel ke DNA template. Protein lain ini berbentuk seperti sliding clamp yang memegang DNA polimerase tetap menempel ke DNA tempate.

Bagaimana cara sliding clamp ini dapat membantu DNA polimerase tetap menempel namun di sisi lain tidak menghalangi enzim ini terdisosiasi saat replikasi sudah selesai?
Kondisi tersebut dapat dipenuhi karena struktur protein memperlihatkan bentuk cincin yang melingkupi DNA double helix. Satu sisi cincin menempel pada bagian belakang DNA polimerase dan sisi lainnya dapat bergeser secara bebas sewaktu DNA polimerase bergeser saat replikasi.
Penyusunan klem di sekitar struktur DNA membutuhkan hidrolisis ATP oleh kompleks protein khusus yaitu clamp loader. Protein ini menghidrolisi ATP agar struktur kompleks ini dapat menempel ke batas primer dan template dari DNA.
Protein-protein di Garpu Repliaksi Bekerja Sama Membentuk Mesin Replikasi
Apabila kita simak penjelasan sejauh ini, tampak bahwa proses replikasi DNA memerlukan banyak protein. Sebenarnya protein ini saling bergabung di garpu replikasi dan menjalankan proses replikasi DNA secara efisien.

Secara ringkas, kerja mesin replikasi ini terdiri dari di bagian depan dari garpu replikasi, enzim DNA helikase membuka struktur heliks DNA. Dua enzim polimerase bekerja bekerja di bagian garpu replikasi, satu di leading strand dan satu lagi di lagging strand. Di leading strand, DNA polimerase bekerja secara kontinu sedangkan di lagging strand DNA polimerase bekerja dengan interval pendek menggunakan RNA primer yang dibuat oleh DNA primase. Kedekatan struktur protein-protein ini meningkatkan efisiensi proses replikasi DNA dan dimungkinkan karena struktur protein penunjang di lagging strand dari garpu replikasi.
Susunan ini juga memungkinkan dapat menempelnya clamp DNA polimerase setiap kali fragmen Okazaki disintesis. Clamp loader dan DNA polimerase di lagging strand dapat tetap berada di tempat walaupun sudah lepas dari template. Mesin repliaksi ini membentuk grup atau unit protein besar dengan ukuran >106 dalton.
Di belakang, mesin replikasi DNA meninggalkan serangkaian fragmen Okazaki yang belum tersambung dan masih mengandung RNA primer. RNA kemudian dibuang dan sistem enzim perbaikan DNA mengganti RNA menjadi DNA dan rangkaian yang belum tersambung diikat oleh enzim DNA ligase.
Sistem Strand-Directed Mismatch Repair Membuang Kesalahan Replikasi yang Lolos dari Mesin Replikasi
Walaupun telah ada mekanisme proofreading eksonuklease yang dijalankan DNA polimerase, kesalahan replikasi DNA masih bisa lolos dari sistem ini. Untuk mengenali dan memperbaiki kesalahan ini ada sistem lain yaitu strand-directed mismatch repair.
Agar sistem ini berjalan secara efektif, maka sistem ini harus mengenali strand yang baru direplikasi dari strand DNA template. Pada E.coli, pada DNA terdapat proses metilasi dari basa A tertentu yakni pada sekuens GATC. Metilasi ini biasanya tidak berlangsung saat itu juga namun menunggu strand yang baru direplikasi untuk selesai diinkorporasikan ke DNA double helix baru.
Oleh sebab itu, strand DNA yang baru tidak punya pola metilasi di posisi A sekuens GATC. Dengan demikian sistem akan mengenali segmen DNA yang baru sehingga mengenali segmen DNA yang salah diduplikasi. Sistem ini terdiri dari 3 langkah yaitu mengenali kesalahan replikasi, mengeluarkan segmen DNA yang mengandung kesalahan, dan membentuk DNA yang baru untuk memperbaiki kesalahan.
Pada manusia, sistem ini juga berlaku. Pada individu yang mengalami defek pada sistem ini memiliki predisposisi atau risiko untuk terkena kanker. Contohnya pada kasus kanker kolorektal yaitu hereditary nonpolyposis colon cancer (HNPCC) dimana apabila terjadi mutasi pada satu gen lain yang fungsional akan mengganggu proses proofreading sehingga mutasi akan berakumulasi dan munculah kanker.
Pada eukariota, mekanisme pengenalan DNA yang baru direplikasi tidak tergantung metilasi DNA. Misalnya pada ragi dan Drosophila, pada kedua organisme ini DNA tidak dimetilasi. Strand DNA yang baru direplikasi biasanya terdapat nick (single DNA break) yang merupakan petanda strand yang baru direplikasi.

DNA Topoisomerase Mencegah Kekusutan DNA Saat Replikasi
Ketika replikasi terus berlangsung, terjadi masalah karena adanya tahanan puntiran dari DNA akibat kerja DNA helikase. Seperti saat kita membuka puntiran tambang, semakin dibuka semakin kuat tahanan yang dirasakan. Begitu pula dengan untaian DNA.

Untuk mengatasi masalah ini, terdapat protein yang dinamakan DNA topoisomerase. Enzim ini merupakan nuklease reversibel yang memecah ikatan fosfodiester dari DNA dan kemudian menyambungkan kembali ikatan tersebut. Terdapat dua tipe topoisomerase yaitu topoisomerase I dan topoisomerase II.
Topoisomerase I
Topoisomerase I akan menyebabkan putusnya sementara satu strand DNA. Putusnya satu bagian dari DNA ini akan memungkinkan rotasi dari untaian DNA. Hal ini akan mengurangi gaya puntiran sehingga replikasi DNA dapat dilanjutkan.
Dikarenakan ikatan kovalen dari protein topoisomerase dengan fosfat DNA mempertahankan tingkat energi yang ada, maka penyambungan kembali strand DNA berlangsung cepat dan tidak membutuhkan energi tambahan.
Topoisomerase II
Tipe kedua dari topoisomerase adalah topoisomerase II. Enzim ini membentuk ikatan kovalen pada kedua strand DNA pada waktu yang bersamaan. Akibatnya, DNA akan terputus secara keseluruhan secara sementara. Enzim ini diaktifkan di kromosom dimana terdapat dua double helix saling bersilangan membentuk simpul.
Saat topoisomerase II bertemu dua DNA yang bersilangan maka dengan menghidrolisis ATP terjadi sebagai berikut: 1) memotong satu double helix secara reversibel membentuk “gerbang” ; 2) menyilangkan double helix kedua melewati gerbang yang dibentuk sebelumnya; 3) menyambung ulang DNA yang sebelumnya diputus.
Dengan kerja topoisomerase II ini, akan mencegah kemungkinan terjadinya kekusutan pada bentang DNA khususnya saat replikasi. Agar lebih jelas mengenai kerja topoisomerase II ini dapat dilihat di gambar di bawah ini:



Kesamaan Proses Replikasi DNA pada Eukariota dan Bakteri
Informasi mengenai proses replikasi ini banyak berasal dari penelitian mengenai replikasi DNA di bakteri dan bakteriofag. Pada dasarnya terdapat banyak kesamaan antara proses replikasi bakteri dengan eukariota. Hanya saja, komponen protein pada mekanisme replikasi eukariota lebih banyak. Sebagai contoh, protein SSB pada eukariota terdiri dari tiga subunit sedangkan pada bakteri hanya satu unit.
Begitu pula dengan DNA primase eukariota yang terdiri dari enzim multisubunit pada eukariota. Kompleks protein ini terdiri dari DNA polimerase yang dinamakan DNA polimerase α-primase. Kompleks protein ini memulai setiap fragmen Okazagi pada lagging strand dengan NRA dan kemudian meneruskan RNA primer dengan segmen pendek DNA. Pada titik ini, dua enzim polimerase replikatif eukariota utama, δ dan ε datang dan melengkapi setiap segmen Okazaki dan secara simultan juga memperpanjang leading strand.